马铃薯晚疫病是全球农业生产中的重大病害,也是植物病理学的模式研究系统之一,曾导致历史上著名的“爱尔兰大饥荒”,至今每年仍造成近百亿美元经济损失(Nature, 2023)。该病害被列入我国《一类农作物病虫害名录》,是严重威胁粮食安全的八大病害之一。由于致病疫霉菌变异迅速,现有抗病基因在育种中逐渐失效,亟需挖掘新型抗性基因资源和抗病育种策略。

近日,我室董莎萌教授课题组联合中国农业科学院深圳农业基因组研究所黄三文团队在《自然》(Nature)期刊发表了题为“Plug-in Strategy for Resistance Engineering Inspired by Potato NLRome”的研究论文,为解决马铃薯晚疫病这一全球性难题提供了一个新方案。
构建马铃薯抗病基因库,揭示抗病基因进化规律

作为全球最重要的非谷物主粮作物,马铃薯生产长期受晚疫病严重制约。尽管科学家已从野生种中克隆出近30个抗晚疫病基因,但多数已被病原菌迅速克服。传统四倍体育种遗传背景复杂,难以实现多抗基因聚合。随着杂交马铃薯育种计划的推进,马铃薯正从无性繁殖的四倍体向种子繁殖的二倍体转变,为抗病育种开辟了新空间。
研究团队选取7个高抗晚疫病的野生马铃薯种质,采用PacBio HiFi高通量测序技术完成高质量基因组组装,完整性高达94.6%–98.3%。结合已公开基因组数据,构建了包含39,211个NLR类抗病基因的“马铃薯泛抗病基因组(Pan-NLRome)”,是目前植物界规模最大、覆盖最全的抗病基因资源库。
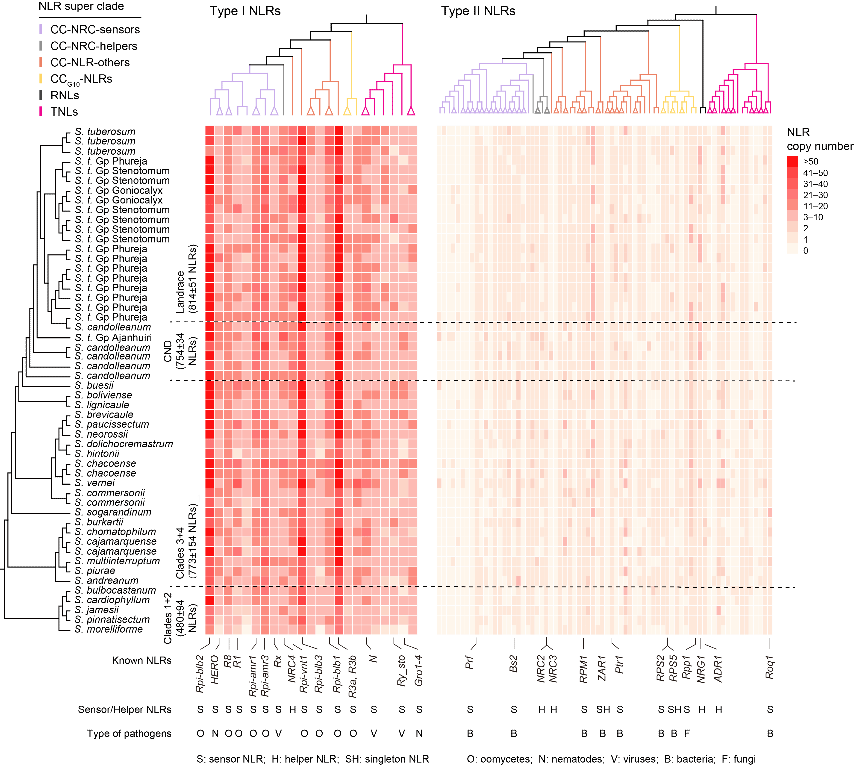
图1.Pan-NLRome中Type I和Type II两类进化特征的抗病基因
研究进一步将抗病基因划分为Type I与Type II两大类:Type I基因拷贝数多、变异快,主要识别卵菌、线虫等快速进化病原;Type II基因保守且数量少,多作为“辅助蛋白”参与免疫信号传递。该发现解释了部分抗病基因易失效的原因,也为功能预测提供了进化依据。
一文实现多基因克隆,为粮食安全注入新资源

基于Pan-NLRome资源,团队成功克隆了三个新型抗晚疫病基因:
Rpi-brk1:通过对马铃薯NLRome中非典型结构域(ID)的进化分析,团队发现HMA结构域在NLR中快速进化,并鉴定到一个携带重金属结合域(HMA)的NLR基因,可直接识别效应蛋白AVRbrk1,其抗病作用依赖HMA结构域与病原菌效应子的直接互作。
Rpi-cjm1:首个在马铃薯中发现的TIR型NLR抗晚疫病基因,能够识别多种AVRblb2家族效应蛋白,抗病谱宽于已知的Rpi-blb2抗病基因。
Rpi-cph1:首次在野生马铃薯S. cardiophyllum中发现,与光果龙葵中已报道的Rpi-amr3同源。
这些抗病新基因为通过常规育种手段精准设计抗晚疫病病性状提供了资源支撑。
创新“Plug-in”育种策略:结构域嫁接拓展抗病谱
更具突破性的是,团队提出“结构域插件(Plug-in)”育种策略:将Rpi-brk1的HMA结构域“嫁接”至已失效的R1蛋白末端,使R1同时识别AVR1与AVRbrk1,成功拓展其抗病谱(图2)。这种“即插即用”的基因设计思路,为快速创制广谱抗病品种提供了全新路径。该策略有望通过基因编辑手段,将功能结构域精准嵌入作物原有抗病基因,绕过转基因壁垒,实现抗性升级。
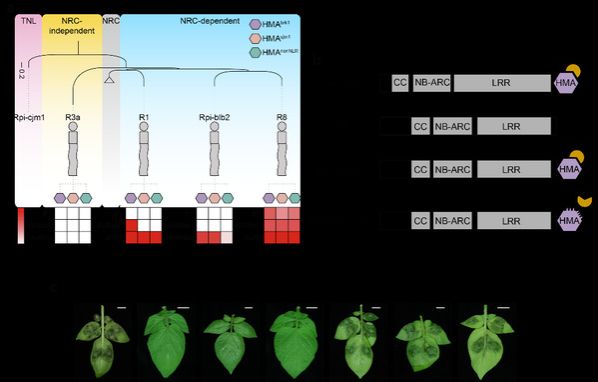
图2. Plug-in抗病育种策略提高马铃薯晚疫病抗性
本研究不仅构建了高质量的抗病基因资源库,建立了作物抗病基因挖掘的基因组学范式,更创新性地提出可推广的育种策略。随着基因编辑技术的不断成熟,“Plug-in”策略有望成为抗病育种的核心工具,助力农作物摆脱与病原体间无休止的“基因军备竞赛”,为全球粮食安全提供持续科技支撑。中国团队打造的这套“免疫外挂”,有望推动作物抗病育种迈入可设计、可预测的新纪元,为保障我国安全,实现农业科技自立自强提供支撑。
我室董莎萌教授与中国农业科学院深圳农业基因组研究所黄三文研究员为本论文共同通讯作者。该研究得到了国家自然科学基金卓越创新群体(B类)、重点项目、广东省基础与应用基础研究重点项目等项目资助。

